Multivariate modelling with minimally biased variable selection (MUVR)
Source:R/multivariate.R
muvr_analysis.RdA wrapper around MUVR2 (random forest,
PLS(-DA)) and MUVR2_EN (elastic net)
functions from the MUVR2 package.
Usage
muvr_analysis(
object,
y = NULL,
id = NULL,
multi_level = FALSE,
multi_level_var = NULL,
covariates = NULL,
static_covariates = NULL,
all_features = FALSE,
nRep = 50,
nOuter = 6,
nInner = nOuter - 1,
varRatio = 0.75,
method = c("PLS", "RF"),
assay.type = NULL,
...
)Arguments
- object
a
SummarizedExperimentorMetaboSetobject- y
character, column name in pheno data of the target variable
- id
character, column name in pheno data of the subject ID variable in case of repeated measurements
- multi_level
logical, whether multi-level modeling should be applied, see Details
- multi_level_var
character, column name in pheno data of the variable for splitting the data in multi-level modeling
- covariates, static_covariates
character, column names of pheno data to use as covariates in the model, in addition to molecular features.
static_covariatesare ignored for non-multi-level models. For multi-level models, the change incovariatesis computed, whilestatic_covariatesare taken from the first time point.- all_features
logical, should all features be included in the model? if FALSE, flagged features are left out
- nRep
Number of repetitions of double CV, parameter of MUVR
- nOuter
Number of outer CV loop segments, parameter of MUVR
- nInner
Number of inner CV loop segments, parameter of MUVR
- varRatio
Ratio of variables to include in subsequent inner loop iteration, parameter of MUVR
- method
Multivariate method. Supports 'PLS', 'RF' and 'EN'
- assay.type
character, assay to be used in case of multiple assays
- ...
other parameters to
MUVR2orMUVR2_ENandgetVar(when method == "EN")
Details
This function is now using the MUVR2 package, characterized as an
upgrade extending the original MUVR package by the inclusion of elastic net
regression (EN) and some functionality not covered by this wrapper. Elastic
net regression supports covariate adjustment by suppressing regularization
of specified features from the regularization procedure. Note that this is
different from simply including covariates such as sex. EN also differs from
PLS and RF in that no recursive variable elimination is performed, so an
additional scheme is used to obtain the 'min', 'mid' and 'max' models using
getVar.
Sex would be entered as a static covariate, since the change in sex is zero for all individuals, so computing the change and using that as a covariate does not make sense.
Note that there are several more plots available in MUVR2 for inspecting the
results, notably plotMV,
plotStability and plotVIRank
Many of these return different plots depending on the model specification.
Examples
data(example_set)
ex_set <- drop_qcs(example_set)[1:10, ]
ex_set$Injection_order %<>% as.numeric()
# Simple PLS regression model
pls_model <- muvr_analysis(ex_set,
y = "Injection_order", nRep = 2, method = "PLS")
#> all 10 variables are numeric.
#> There are originally 10 variables
#> They are transformed into 10 variables by onehotencoding
#> X is transformed to a matrix by onehotencoding.
#> Warning:
#> Missing ID -> Assume all unique (i.e. sample independence)
#>
#> Missing fitness -> RMSEP
#> Warning: executing %dopar% sequentially: no parallel backend registered
#> Type 'citation("pROC")' for a citation.
#>
#> Attaching package: ‘pROC’
#> The following objects are masked from ‘package:IRanges’:
#>
#> cov, var
#> The following objects are masked from ‘package:S4Vectors’:
#>
#> cov, var
#> The following object is masked from ‘package:BiocGenerics’:
#>
#> var
#> The following objects are masked from ‘package:stats’:
#>
#> cov, smooth, var
#>
#> Repetition 1 of 2:
#>
#> Segment 1 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 2 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 3 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 4 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 5 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 6 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Repetition 2 of 2:
#>
#> Segment 1 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 2 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 3 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 4 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 5 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 6 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Elapsed time 0.0406833333333334 mins
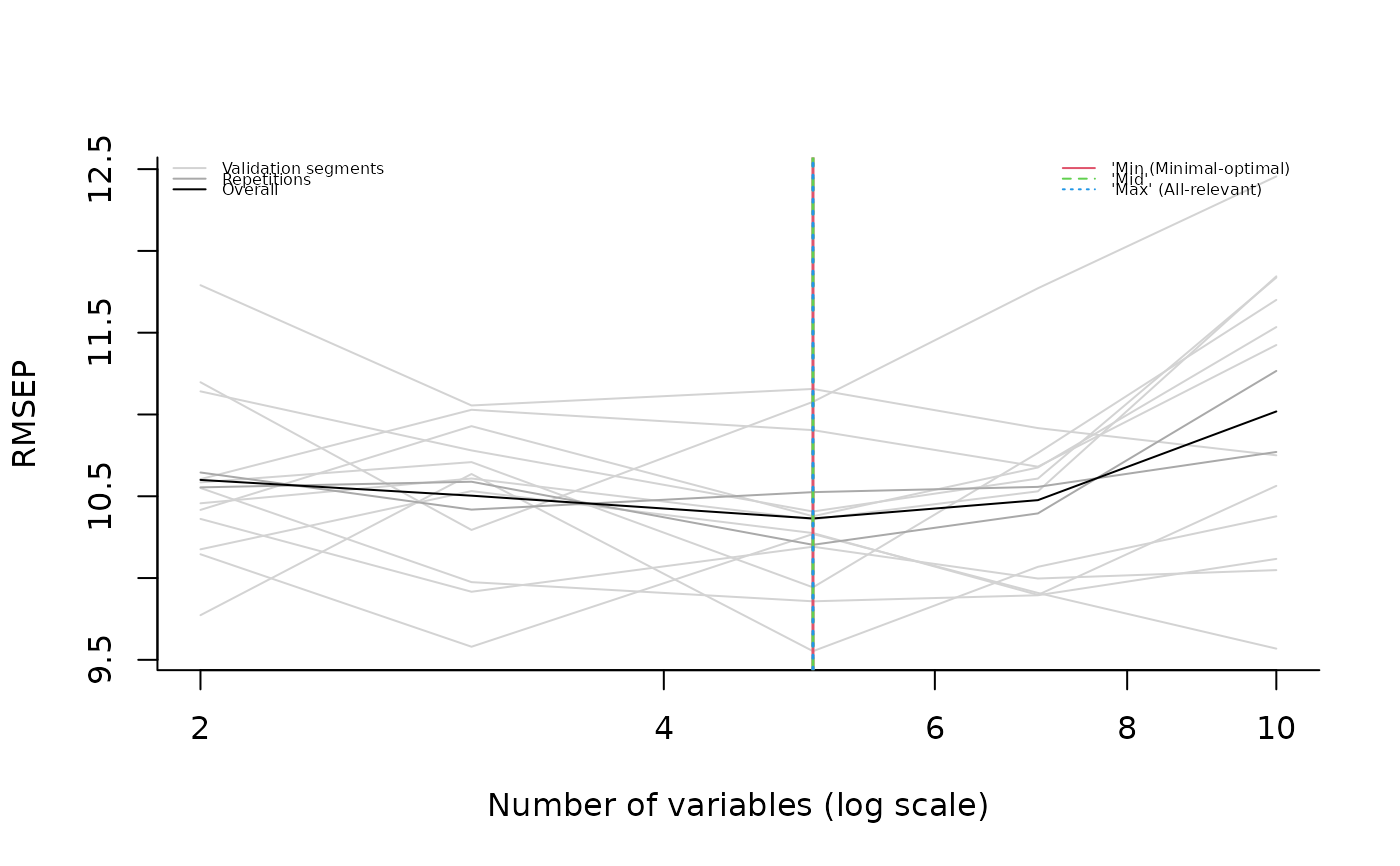 # RF classification with covariate and repeated measures (not longitudinal)
rf_model <- muvr_analysis(ex_set, y = "Group", id = "Subject_ID",
nRep = 2, method = "RF", covariates = "Injection_order")
#>
#> Y is factor -> Classification (2 classes)
#>
#> Missing fitness -> BER
#> randomForest 4.7-1.2
#> Type rfNews() to see new features/changes/bug fixes.
#>
#> Attaching package: ‘randomForest’
#> The following object is masked from ‘package:ggplot2’:
#>
#> margin
#> The following object is masked from ‘package:Biobase’:
#>
#> combine
#> The following object is masked from ‘package:BiocGenerics’:
#>
#> combine
#>
#> Repetition 1 of 2:
#>
#> Segment 1 (variables):
#> 11
#> .
#> .
#> .
#> .
#> .
#> 8
#> .
#> .
#> .
#> .
#> .
#> 6
#> .
#> .
#> .
#> .
#> .
#> 4
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 2 (variables):
#> 11
#> .
#> .
#> .
#> .
#> .
#> 8
#> .
#> .
#> .
#> .
#> .
#> 6
#> .
#> .
#> .
#> .
#> .
#> 4
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 3 (variables):
#> 11
#> .
#> .
#> .
#> .
#> .
#> 8
#> .
#> .
#> .
#> .
#> .
#> 6
#> .
#> .
#> .
#> .
#> .
#> 4
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 4 (variables):
#> 11
#> .
#> .
#> .
#> .
#> .
#> 8
#> .
#> .
#> .
#> .
#> .
#> 6
#> .
#> .
#> .
#> .
#> .
#> 4
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 5 (variables):
#> 11
#> .
#> .
#> .
#> .
#> .
#> 8
#> .
#> .
#> .
#> .
#> .
#> 6
#> .
#> .
#> .
#> .
#> .
#> 4
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 6 (variables):
#> 11
#> .
#> .
#> .
#> .
#> .
#> 8
#> .
#> .
#> .
#> .
#> .
#> 6
#> .
#> .
#> .
#> .
#> .
#> 4
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Repetition 2 of 2:
#>
#> Segment 1 (variables):
#> 11
#> .
#> .
#> .
#> .
#> .
#> 8
#> .
#> .
#> .
#> .
#> .
#> 6
#> .
#> .
#> .
#> .
#> .
#> 4
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 2 (variables):
#> 11
#> .
#> .
#> .
#> .
#> .
#> 8
#> .
#> .
#> .
#> .
#> .
#> 6
#> .
#> .
#> .
#> .
#> .
#> 4
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 3 (variables):
#> 11
#> .
#> .
#> .
#> .
#> .
#> 8
#> .
#> .
#> .
#> .
#> .
#> 6
#> .
#> .
#> .
#> .
#> .
#> 4
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 4 (variables):
#> 11
#> .
#> .
#> .
#> .
#> .
#> 8
#> .
#> .
#> .
#> .
#> .
#> 6
#> .
#> .
#> .
#> .
#> .
#> 4
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 5 (variables):
#> 11
#> .
#> .
#> .
#> .
#> .
#> 8
#> .
#> .
#> .
#> .
#> .
#> 6
#> .
#> .
#> .
#> .
#> .
#> 4
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 6 (variables):
#> 11
#> .
#> .
#> .
#> .
#> .
#> 8
#> .
#> .
#> .
#> .
#> .
#> 6
#> .
#> .
#> .
#> .
#> .
#> 4
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Elapsed time 0.03065 mins
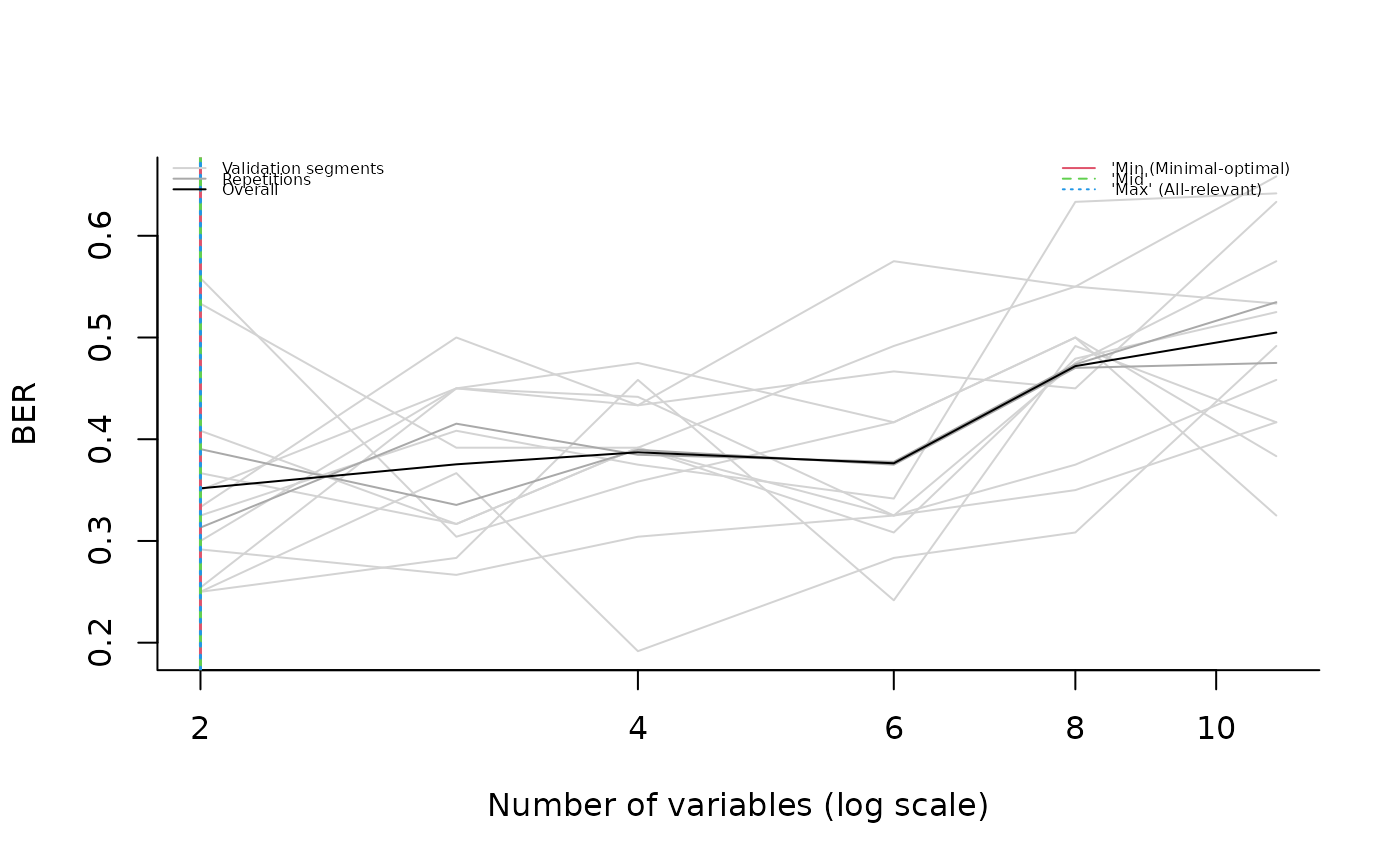
# RF classification with covariate and repeated measures (not longitudinal)
rf_model <- muvr_analysis(ex_set, y = "Group", id = "Subject_ID",
nRep = 2, method = "RF", covariates = "Injection_order")
#>
#> Y is factor -> Classification (2 classes)
#>
#> Missing fitness -> BER
#> randomForest 4.7-1.2
#> Type rfNews() to see new features/changes/bug fixes.
#>
#> Attaching package: ‘randomForest’
#> The following object is masked from ‘package:ggplot2’:
#>
#> margin
#> The following object is masked from ‘package:Biobase’:
#>
#> combine
#> The following object is masked from ‘package:BiocGenerics’:
#>
#> combine
#>
#> Repetition 1 of 2:
#>
#> Segment 1 (variables):
#> 11
#> .
#> .
#> .
#> .
#> .
#> 8
#> .
#> .
#> .
#> .
#> .
#> 6
#> .
#> .
#> .
#> .
#> .
#> 4
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 2 (variables):
#> 11
#> .
#> .
#> .
#> .
#> .
#> 8
#> .
#> .
#> .
#> .
#> .
#> 6
#> .
#> .
#> .
#> .
#> .
#> 4
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 3 (variables):
#> 11
#> .
#> .
#> .
#> .
#> .
#> 8
#> .
#> .
#> .
#> .
#> .
#> 6
#> .
#> .
#> .
#> .
#> .
#> 4
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 4 (variables):
#> 11
#> .
#> .
#> .
#> .
#> .
#> 8
#> .
#> .
#> .
#> .
#> .
#> 6
#> .
#> .
#> .
#> .
#> .
#> 4
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 5 (variables):
#> 11
#> .
#> .
#> .
#> .
#> .
#> 8
#> .
#> .
#> .
#> .
#> .
#> 6
#> .
#> .
#> .
#> .
#> .
#> 4
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 6 (variables):
#> 11
#> .
#> .
#> .
#> .
#> .
#> 8
#> .
#> .
#> .
#> .
#> .
#> 6
#> .
#> .
#> .
#> .
#> .
#> 4
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Repetition 2 of 2:
#>
#> Segment 1 (variables):
#> 11
#> .
#> .
#> .
#> .
#> .
#> 8
#> .
#> .
#> .
#> .
#> .
#> 6
#> .
#> .
#> .
#> .
#> .
#> 4
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 2 (variables):
#> 11
#> .
#> .
#> .
#> .
#> .
#> 8
#> .
#> .
#> .
#> .
#> .
#> 6
#> .
#> .
#> .
#> .
#> .
#> 4
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 3 (variables):
#> 11
#> .
#> .
#> .
#> .
#> .
#> 8
#> .
#> .
#> .
#> .
#> .
#> 6
#> .
#> .
#> .
#> .
#> .
#> 4
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 4 (variables):
#> 11
#> .
#> .
#> .
#> .
#> .
#> 8
#> .
#> .
#> .
#> .
#> .
#> 6
#> .
#> .
#> .
#> .
#> .
#> 4
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 5 (variables):
#> 11
#> .
#> .
#> .
#> .
#> .
#> 8
#> .
#> .
#> .
#> .
#> .
#> 6
#> .
#> .
#> .
#> .
#> .
#> 4
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 6 (variables):
#> 11
#> .
#> .
#> .
#> .
#> .
#> 8
#> .
#> .
#> .
#> .
#> .
#> 6
#> .
#> .
#> .
#> .
#> .
#> 4
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Elapsed time 0.03065 mins
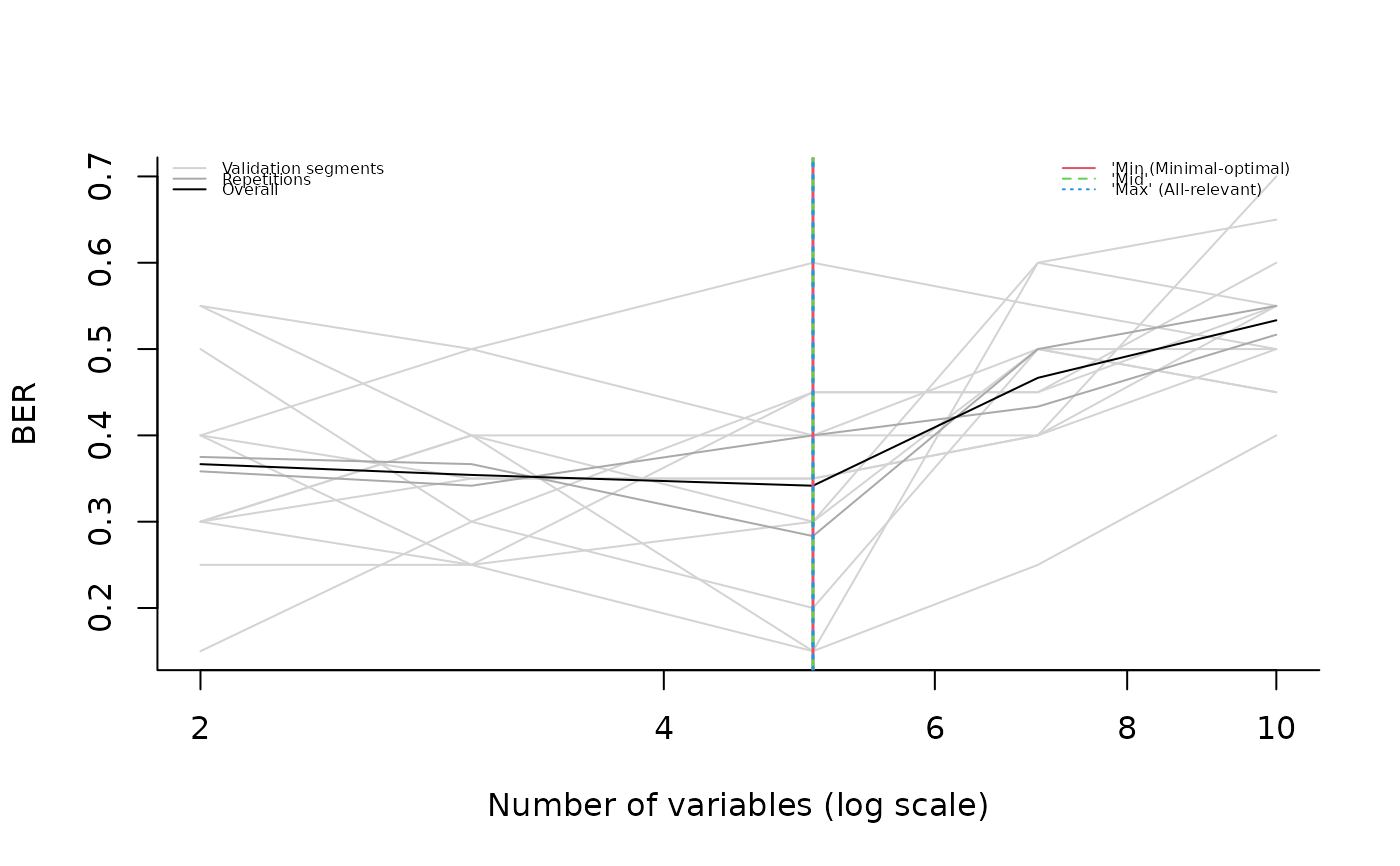
 # RF classification on multilevel variable comparing levels of y
rf_model_ <- muvr_analysis(ex_set,
y = "Group", multi_level = TRUE, id = "Subject_ID",
multi_level_var = "Time", method = "RF", nRep = 2)
#> Computing effect matrix according to Time : 2 - 1
#> Warning:
#> Missing ID -> Assume all unique (i.e. sample independence)
#>
#> Y is factor -> Classification (2 classes)
#>
#> Missing fitness -> BER
#>
#> Repetition 1 of 2:
#>
#> Segment 1 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 2 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 3 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 4 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 5 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 6 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Repetition 2 of 2:
#>
#> Segment 1 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 2 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 3 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 4 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 5 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 6 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Elapsed time 0.0216166666666667 mins
# RF classification on multilevel variable comparing levels of y
rf_model_ <- muvr_analysis(ex_set,
y = "Group", multi_level = TRUE, id = "Subject_ID",
multi_level_var = "Time", method = "RF", nRep = 2)
#> Computing effect matrix according to Time : 2 - 1
#> Warning:
#> Missing ID -> Assume all unique (i.e. sample independence)
#>
#> Y is factor -> Classification (2 classes)
#>
#> Missing fitness -> BER
#>
#> Repetition 1 of 2:
#>
#> Segment 1 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 2 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 3 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 4 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 5 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 6 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Repetition 2 of 2:
#>
#> Segment 1 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 2 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 3 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 4 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 5 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Segment 6 (variables):
#> 10
#> .
#> .
#> .
#> .
#> .
#> 7
#> .
#> .
#> .
#> .
#> .
#> 5
#> .
#> .
#> .
#> .
#> .
#> 3
#> .
#> .
#> .
#> .
#> .
#> 2
#> .
#> .
#> .
#> .
#> .
#>
#> Elapsed time 0.0216166666666667 mins
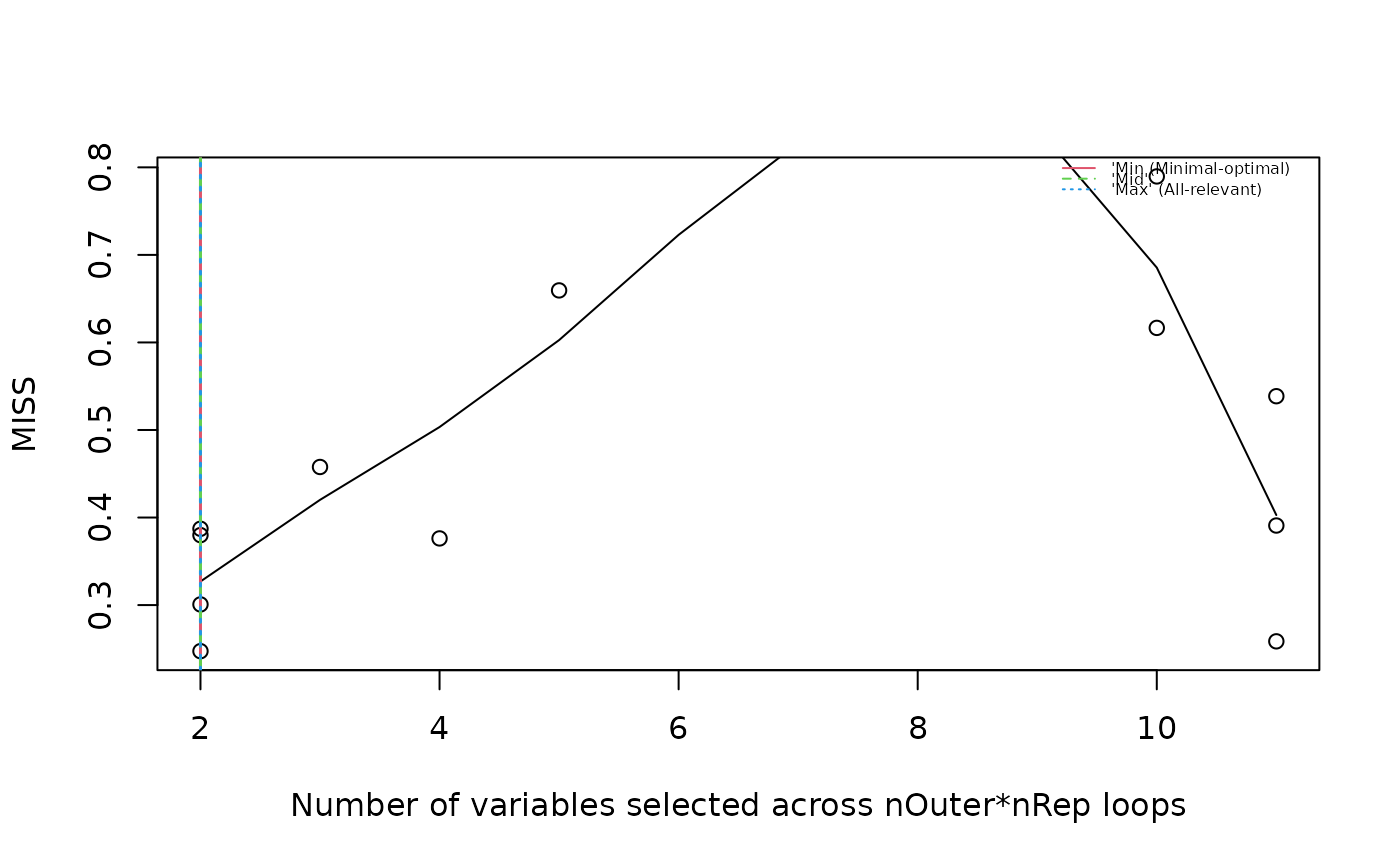
 # EN regression on multilevel variable with covariate and static covariate
ex_set$Group %<>% as.numeric()
en_model <- muvr_analysis(ex_set, id = "Subject_ID",
multi_level = TRUE, multi_level_var = "Time",
covariates = "Injection_order", static_covariates = "Group",
method = "EN", nRep = 2)
#> Computing effect matrix according to Time : 2 - 1
#> all 12 variables are numeric.
#> There are originally 12 variables
#> They are transformed into 12 variables by onehotencoding
#> X is transformed to a matrix by onehotencoding.
#>
#> 1variables with near zero variance detected -> removed from X and stored under $nzv
#>
#> Missing ID -> Assume all unique (i.e. sample independence)
#>
#> Multilevel -> Regression on (-1,1) & fitness=MISS
#> Loading required package: Matrix
#>
#> Attaching package: ‘Matrix’
#> The following object is masked from ‘package:S4Vectors’:
#>
#> expand
#> Loaded glmnet 4.1-9
#>
#> Repetition 1 of 2:
#>
#> Segment 1 (Inner repeat):
#> 1...
#>
#> Segment 2 (Inner repeat):
#> 1...
#>
#> Segment 3 (Inner repeat):
#> 1...
#>
#> Segment 4 (Inner repeat):
#> 1...
#>
#> Segment 5 (Inner repeat):
#> 1...
#>
#> Segment 6 (Inner repeat):
#> 1...
#>
#> Repetition 2 of 2:
#>
#> Segment 1 (Inner repeat):
#> 1...
#>
#> Segment 2 (Inner repeat):
#> 1...
#>
#> Segment 3 (Inner repeat):
#> 1...
#>
#> Segment 4 (Inner repeat):
#> 1...
#>
#> Segment 5 (Inner repeat):
#> 1...
#>
#> Segment 6 (Inner repeat):
#> 1...
#>
#> Elapsed time 0.0613000000000001 mins
# EN regression on multilevel variable with covariate and static covariate
ex_set$Group %<>% as.numeric()
en_model <- muvr_analysis(ex_set, id = "Subject_ID",
multi_level = TRUE, multi_level_var = "Time",
covariates = "Injection_order", static_covariates = "Group",
method = "EN", nRep = 2)
#> Computing effect matrix according to Time : 2 - 1
#> all 12 variables are numeric.
#> There are originally 12 variables
#> They are transformed into 12 variables by onehotencoding
#> X is transformed to a matrix by onehotencoding.
#>
#> 1variables with near zero variance detected -> removed from X and stored under $nzv
#>
#> Missing ID -> Assume all unique (i.e. sample independence)
#>
#> Multilevel -> Regression on (-1,1) & fitness=MISS
#> Loading required package: Matrix
#>
#> Attaching package: ‘Matrix’
#> The following object is masked from ‘package:S4Vectors’:
#>
#> expand
#> Loaded glmnet 4.1-9
#>
#> Repetition 1 of 2:
#>
#> Segment 1 (Inner repeat):
#> 1...
#>
#> Segment 2 (Inner repeat):
#> 1...
#>
#> Segment 3 (Inner repeat):
#> 1...
#>
#> Segment 4 (Inner repeat):
#> 1...
#>
#> Segment 5 (Inner repeat):
#> 1...
#>
#> Segment 6 (Inner repeat):
#> 1...
#>
#> Repetition 2 of 2:
#>
#> Segment 1 (Inner repeat):
#> 1...
#>
#> Segment 2 (Inner repeat):
#> 1...
#>
#> Segment 3 (Inner repeat):
#> 1...
#>
#> Segment 4 (Inner repeat):
#> 1...
#>
#> Segment 5 (Inner repeat):
#> 1...
#>
#> Segment 6 (Inner repeat):
#> 1...
#>
#> Elapsed time 0.0613000000000001 mins